Le big data à la recherche de nouvelles molécules naturelles bioactives
Petit à petit, le big data, ou grand jeux de données, se répand dans toutes les sciences et la biologie moléculaire n’y fait pas exception. Plusieurs nouvelles molécules, inhibitrices d’une protéine jouant un rôle dans le développement de certains cancers, viennent d'être découvertes par l'Institut de chimie des substances naturelles grâce à l’emploi des "réseaux moléculaires", une méthode bio-informatique capable de dénicher de nouvelles molécules d'intérêt à partir de l’analyse simultanée de plusieurs centaines d’extraits naturels. Grâce au protocole expérimental développé par Marc Litaudon et ses collègues, il a été possible de révéler ces molécules bioactives au sein d’un mélange d’extraits contenant chacun plusieurs centaines de molécules.
La nature recèle encore une infinité de secrets inexplorés. Une chance mais aussi un problème car nombreux sont ces secrets qui se ressemblent comme deux gouttes d'eau. Ainsi, il n'est pas rare de "redécouvrir" une molécule déjà connue. Pour ne pas tomber deux fois sur la même aiguille dans une botte de foin, les chimistes de l’Institut de chimie des substances naturelles, à Gif-sur-Yvette, ont sélectionné 300 extraits provenant de plantes de la famille des Euphorbiaceae, endémiques de Nouvelle-Calédonie pour la plupart. Ces extraits ont ensuite été analysé par spectrométrie de masse (LC/MS2), une technique qui classe les molécules en fonction de leur rapport masse/charge. C'est à partir de là que le "Big Data" entre en jeu. Les chimistes se muent alors en détectives informatiques.
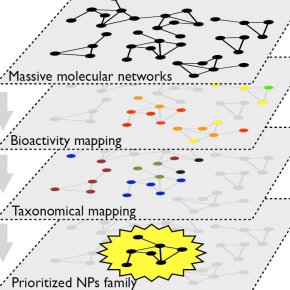
« La première étape consiste à regrouper ces molécules en fonction de leur similarité structurale, détaille l'ingénieur de recherche Marc Litaudon. Cette étape est uniquement informatique. À partir de l’utilisation d’un logiciel en accès libre sur le site « Global natural product social molecular networking » (GNPS http://gnps.ucsd.edu) nous regroupons les molécules selon leur structure pour les représenter dans un espace à 2 dimensions. Un peu comme si on reconstituait les constellations dans un ciel étoilé. » Ces constellations forment les fameux "réseaux moléculaires" qui représentent l’ensemble du contenu moléculaire des espèces étudiées. Une fois les molécules organisées, les chimistes commencent l'enquête. L’application de différents filtres, biologique, taxonomique ou autres, permet alors de faire ressortir les constellations d’intérêt. Il ne reste plus qu’à sélectionner et isoler les perles rares. Quatre perles pour plusieurs milliers de possibilités dans le cas d’une première publication parue le 22 août 2017 dans les pages d’ACS Chemical Biology, suivi de la découverte de 5 autres molécules originales dans une deuxième publication parue le 7 juin 2018 (Org. Chem. Frontiers). D’autres publications sont en cours.
©Marc Litaudon