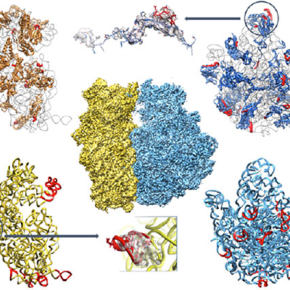
Examen à haute résolution de la machine de synthèse des protéines du staphylocoque doré
Des équipes de l’Institut de biologie moléculaire et cellulaire et de l’Institut de génétique et de biologie moléculaire et cellulaire, ont résolu la structure du ribosome de Staphylococcus aureus par cryomicroscopie électronique. Vue de loin, l’architecture de son usine à fabriquer des protéines est largement similaire à celle de bactéries non pathogènes. Mais vue de près, grâce à ces travaux, elle révèle des disparités suggérant des variantes au mécanisme de synthèse des protéines chez les bactéries qui seraient propres au staphylocoque doré. Ces différences pourraient représenter autant de nouvelles cibles potentielles pour des thérapies sélectives de ce redoutable pathogène. Cette étude est publiée dans la revue Nucleic Acids Research.
Selon l’Institut national de la santé et de la recherche médicale (Inserm), un patient sur vingt contracte une infection lors d’un séjour à l’hôpital en France, en particulier dans le cas d’interventions invasives. Environ la moitié des 750 000 infections dites ‘nosocomiales’ que cela représente chaque année, est due à une bactérie, principalement Escherichia coli (26%), Staphylococcus aureus (16%) ou Pseudomonas aeruginosa (8%). Dans 20 à 50% des cas, le germe qui provoque l’infection est résistant à tout traitement antibiotique connu à ce jour, ce qui est source d’une crise médicale sans précédent depuis l’avènement des antibiotiques au cours de la première moitié du siècle dernier. Cette situation alarmante stimule le développement de nouveaux médicaments et la recherche de nouvelles stratégies pour combattre ces infections.
Un projet de recherche pluridisciplinaire et collaboratif a été élaboré par plusieurs équipes de l’Institut de Biologie Moléculaire et Cellulaire et de l’Institut de Génétique et de Biologie Moléculaire et Cellulaire afin de caractériser le mode de fonctionnement du staphylocoque doré. C’est dans ce cadre qu’a été déterminée la structure fine de la machine de synthèse des protéines de S. aureus, le ribosome.
Le ribosome de S. aureus ressemble à celui d’autres bactéries : il possède deux sous-unités de tailles différentes, que composent environ cinquante protéines organisées dans l’espace par quelques chaînes d’acides nucléiques longues de plusieurs centaines de résidus. L’ensemble représente plus de 140 000 atomes qui ont pu être modélisés en trois dimensions grâce à la puissance actuelle des techniques de microscopie, d’analyse d’image, et de reconstruction graphique.
Le staphylocoque doré étant une bactérie, il n’est pas surprenant de noter que son ribosome s’organise comme ceux déjà connus d’autres bactéries. Globalement, il possède les mêmes éléments nécessaires à la synthèse des protéines, aux mêmes endroits de sa structure, tels que par exemple les sites de fixation des adaptateurs qui apportent les acides aminés et des facteurs annexes qui interviennent à chaque cycle d’incorporation de ces acides aminés dans une protéine en voie de synthèse. Cependant, une comparaison structurale plus fine des ribosomes de plusieurs bactéries montre des différences (voir figure). En particulier, les différences structurales observées pour une protéine (bL31) et une hélice d’acide ribonucléique (hélice 26) suggèrent des mécanismes de régulation de la synthèse des protéines propres à S. aureus.
Le ribosome étant la cible de plus de la moitié des antibiotiques connus, cette découverte n’est pas sans conséquence pour la sphère biomédicale. En effet, la carte tridimensionnelle délivrée par ces travaux devrait permettre de guider les expérimentations fonctionnelles qui conduiront à terme à l’identification de mécanismes cellulaires propres au staphylocoque doré, et on peut l’espérer à ses souches résistantes. Ces efforts donneront lieu à l’élaboration de stratégies thérapeutiques, basées sur l’antibiothérapie ou non, spécifiques de ce pathogène.
Même si la signification de toutes les différences structurales entre ribosomes bactériens nous échappe encore, les résultats de ces équipes strasbourgeoises illustrent que tout détail peut avoir son importance. C’est bien dans ces détails de structure entre ribosomes que se trouvent les clés qui permettront un jour de combattre les agents infectieux comme S. aureus avec une plus grande sélectivité, tout en évitant les effets secondaires dus par exemple au déséquilibre de la flore intestinale que provoque les antibiotiques à large spectre.
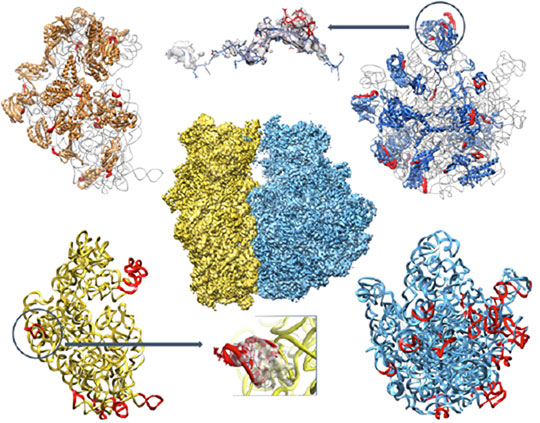
© Quentin Vicens, Yaser Hashem, Iskander Khusainov, Marat Yusupov
En savoir plus
-
Structure of the 70S ribosome from human pathogen Staphylococcus aureus.
Khusainov I, Vicens Q, Bochler A, Grosse F, Myasnikov A, Ménétret J-F, Chicher J, Marzi S, Romby P, Yusupova G, Yusupov M, Hashem Y.
Nucl. Acids Res. October 18, 2016 doi:10.1093/nar/gkw933