Structure et histoire évolutive d’une arbalète bactérienne
Certaines bactéries, en particulier les pathogènes, sont armées d’un système qui fonctionne comme une arbalète et qui leur permet d’éliminer les autres microbes avec lesquelles elles sont en compétition. Elles peuvent aussi s’en servir pour attaquer des cellules eucaryotes telles que amibes et macrophages. Les équipes d’Eric Cascales au laboratoire d’Ingénierie des systèmes macromoléculaires et de Christian Cambillau et Alain Roussel au laboratoire Architecture et fonction des macromolécules biologiques, révèlent la structure d’un composant permettant l’ancrage et l’assemblage de la flèche de l’arbalète. Cette étude a été publiée le 26 juin 2017 dans la revue Nature Microbiology.
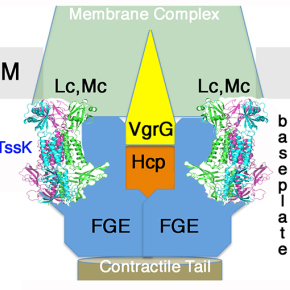
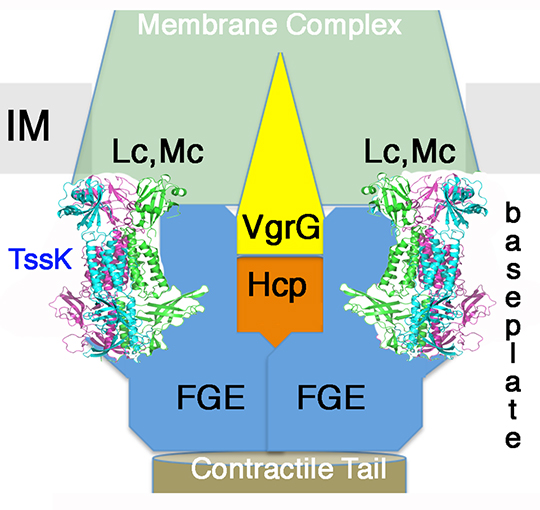
Dans l’environnement ou dans le corps humain, les bactéries ne vivent pas seules. Elles sont en contact avec d’autres populations bactériennes, des organismes unicellulaires ou des cellules eucaryotes. Pour mieux coloniser l’environnement, les bactéries ont développé, parmi d’autres, des mécanismes antagonistes pour pouvoir éliminer les autres espèces bactériennes et faciliter leur accès aux ressources. Parmi ceux mis en jeu pour tuer les autres bactéries, le plus efficace est le système de sécrétion de Type VI (T6SS), composé d’une structure contractile qui fonctionne à l’instar d’une micro-arbalète, et qui ressemble aux virus infectant les bactéries, les bactériophages. La flèche de l’arbalète, en particulier, ressemble à la tige contractile de certains de ces virus, faisant penser que le T6SS est un bactériophage capturé et « retourné » contre les bactéries ennemies au cours de l'évolution. La flèche de l’arbalète se termine par une pointe recouverte de toxines. Elle est entourée par un fourreau agissant comme un ressort dont la contraction propulse la flèche en direction de la bactérie cible pour la tuer. Cette structure synthétisée dans la cellule prédatrice est ancrée à sa membrane par un complexe membranaire multiprotéique. Une structure appelée la plaque basale se fixe au complexe membranaire et permet l’ancrage et l’assemblage de la flèche de l’arbalète.
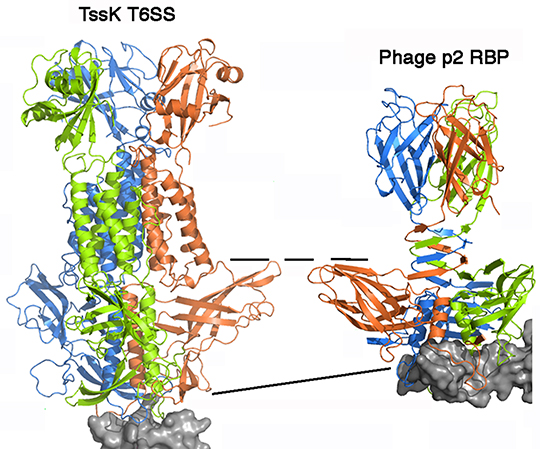
Dans cette nouvelle étude, ces chercheurs ont pu déterminer, par diffraction des rayons X, la structure et le mode d’attachement d’un composant essentiel de la plaque basale (TssK). Van Son Nguyen de l’équipe Architecture et Fonction des Macromolécules Biologiques, s’est servi de domaines d’anticorps de Lama, appelés nanobodies, pour obtenir des cristaux de la molécule TssK utilisables pour déchiffrer sa structure tridimensionnelle. Ces travaux permettent non seulement de savoir comment s’assemble la structure TssK, mais ils apportent également des informations concernant l’origine de ce composant qui provient d’un autre groupe de bactériophages. Ces résultats révèlent l’existence d’un processus d’évolution complexe du T6SS, dans lequel des éléments provenant de différents virus bactériens ont été assemblés.
© Christian Cambillau
© Christian Cambillau
En savoir plus
-
Type VI secretion TssK baseplate protein exhibits structural similarity with phage receptor-binding proteins and evolved to bind the membrane complex.
Nguyen VS, Logger L, Spinelli S, Legrand P, Huyen Pham TT, Nhung Trinh TT, Cherrak Y, Zoued A, Desmyter A, Durand E, Roussel A, Kellenberger C, Cascales E, Cambillau C.
Nat Microbiol. 2017 Jun 26;2:17103. doi: 10.1038/nmicrobiol.2017.103.