Micro-algues marines : maxi-diversité génétique
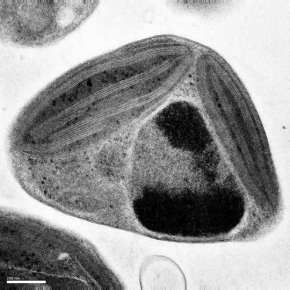
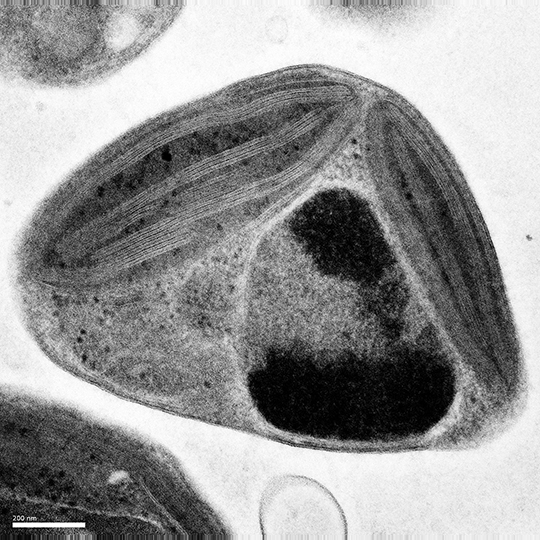
Les microalgues planctoniques, invisibles à l’œil nu, sont à la base de la chaine alimentaire de l’écosystème marin et constituent un réservoir de biodiversité encore méconnu. L’analyse des génomes complet d’une population naturelle d’une minuscule microalgue verte, Ostreococcus tauri, par un consortium international a permis de révéler une hypervariabilité chromosomique inédite, associée à l’immunité cellulaire aux virus à ADN double brin. Cette étude a été publiée le 5 juillet 2017 dans la revue Science Advances.
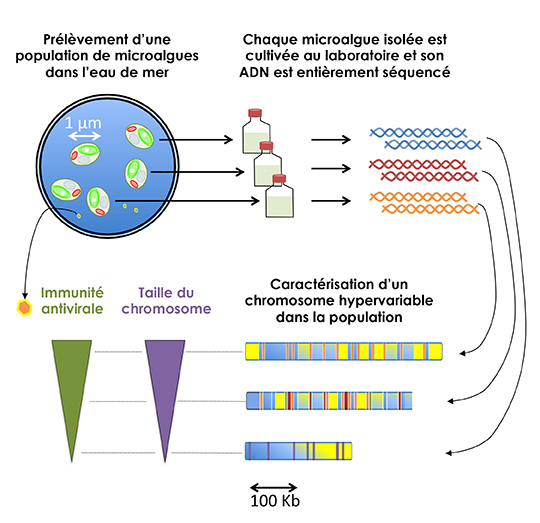
Un consortium international composé de chercheurs de trois laboratoires Français 1 , d’un centre de séquençage américain 2 et d’un laboratoire du Royaume-Uni 3 , coordonné par l’équipe « Genomique Evolutive et Environnementale du Phytoplankton » au laboratoire de Biologie Intégrative des Organismes Marins, a caractérisé la diversité génétique d’une population naturelle de la microalgue verte, Ostreococcustauri (Chlorophyte) échantillonnée dans le Golfe du Lion.
L’utilisation des nouvelles technologies de séquençage « long reads » (PacBio) a permis aux chercheurs de résoudre non seulement l’énigmatique séquence des chromosomes portant le locus du déterminisme sexuel de cette microalgue, mais aussi la structure d’un chromosome impliqué dans l’immunité de ces microalgues aux virus à ADN double brin omniprésents dans leur milieu naturel. Ce chromosome présente une hypervariabilité génétique caractérisée par des réarrangements massifs, accompagnés de duplications en série de petites séquences de ce chromosome, ainsi que d’intégrations de séquences d’ADN extra-chromosomiques.
Ce type de réarrangements massifs du patrimoine génétique n’est pas sans rappeler le phénomène de chromothripsis observé récemment dans les génomes de cellules cancéreuses. Il est encore trop tôt pour établir un lien entre infection virale, réarrangements chromosomiques et immunité cellulaire mais l’élucidation des mécanismes moléculaires à l’origine de cette hypervariabilité génétique devrait permettre d’apporter des éléments de réponse à cette question. Cette analyse suggère un rôle insoupçonné des virus à ADN double brin dans l’évolution de l’architecture des génomes eucaryotes.
© Martin Hohmann-Marriott
© Equipe de Génomique Evolutive et Environnementale du Phytoplancton (GENOPHY).
En savoir plus
-
Population genomics of picophytoplankton unveils novel chromosome hypervariability.
Blanc-Mathieu R, Krasovec M, Hebrard M, Yau S, Desgranges E, Martin J, Schackwitz W, Kuo A, Salin G, Donnadieu C, Desdevises Y, Sanchez-Ferandin S, Moreau H, Rivals E, Grigoriev IV, Grimsley N, Eyre-Walker A, Piganeau G.
Sci Adv. 2017 Jul 5;3(7):e1700239. doi: 10.1126/sciadv.1700239. eCollection 2017 Jul.