Un nouveau mécanisme d'activation de l'expression des gènes chez Mycobacterium tuberculosis
Chez les bactéries, la sous-unité sigma de l'ARN polymérase contrôle la reconnaissance des promoteurs et l'initiation de la synthèse de l'ARN. En utilisant la méthode de transfert d'énergie de Förster entre molécules uniques (smFRET), les chercheurs ont démontré que Mycobacterium tuberculosis utilise un mécanisme d'activation transcriptionnelle encore inconnu dans lequel l'ARN polymérase conserve la sous-unité sigma en conformation inactive "fermée" sauf si elle est stabilisée en conformation active "ouverte" par la protéine activatrice RbpA. Cette étude a été publiée le 23 mai 2018 dans la revue ScienceAdvances
Mycobacterium tuberculosis, l'agent pathogène humain causant la tuberculose, est responsable de près de 1,5 millions de décès annuels le monde. Environ 5000 nouveaux cas sont diagnostiqués chaque année en France. Le développement de nouvelles stratégies thérapeutiques requiert une connaissance approfondie des mécanismes spécifiques de la régulation des gènes permettant à M. tuberculosis de survivre aux traitements par les antibiotiques et de provoquer une infection récurrente.
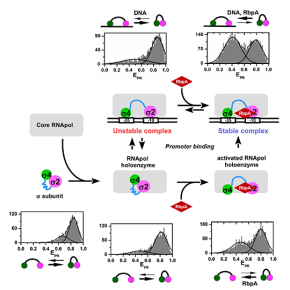
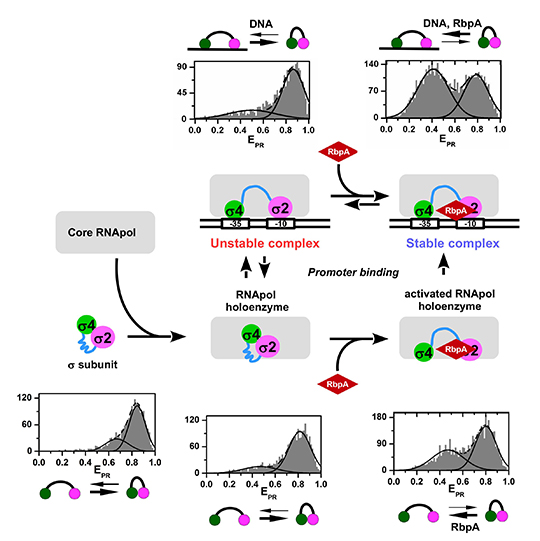
La première étape de l'expression des gènes, l'initiation de la transcription, est réalisée chez les bactéries par l'ARN polymérase ADN-dépendante (l'ARNP), un complexe enzymatique composée de plusieurs sous-unités. La sous-unité dissociable "sigma" de l'ARNP contrôle la reconnaissance des promoteurs et l'initiation de la synthèse de l'ARN. La sous-unité sigma libre en solution adopte une conformation "fermée", inactive, incompatible avec sa liaison au promoteur. Le modèle conventionnel de l'activation de sigma suggère que sa fixation à l'ARNP induit sa transition vers une conformation active "ouverte" optimale pour la reconnaissance du promoteur.
En utilisant la méthode de transfert d'énergie de Förster entre molécules uniques (smFRET), les chercheurs démontré que M. tuberculosis utilise un mécanisme d'activation encore inconnu dans lequel l'ARNP conserve la sous-unité sigma en conformation "fermée" sauf si elle est stabilisée en conformation "ouverte" par la protéine activatrice mycobactérienne RbpA .
Ces résultats suggèrent que le contrôle de la dynamique conformationnelle de la sous-unité sigma par RbpA joue un rôle central dans la régulation de l'expression des gènes chez M. tuberculosis. RbpA est une protéine essentielle impliquée dans la résistance aux antibiotiques et dans le contrôle de la croissance bactérienne. De ce fait, ces résultats peuvent avoir des implications majeures pour comprendre les mécanismes de pathogenèse et envisager de nouvelles approches thérapeutiques.
© Konstantin Brodolin & Emmanuel Margeat
En savoir plus
-
Single-molecule analysis reveals the mechanism of transcription activation in M. tuberculosis.
Vishwakarma RK, Cao AM, Morichaud Z, Perumal AS, Margeat E, Brodolin K.
Sci Adv. 2018 May 23;4(5):eaao5498. doi: 10.1126/sciadv.aao5498. eCollection 2018 May.