Un facteur d’empaquetage de l’ADN paternel est aussi responsable d’un cancer agressif
Pour transmettre le génome du père, les spermatozoïdes doivent quitter l’organisme producteur et nager jusqu’à l’œuf dans un environnement hostile. Une équipe de chercheurs vient de décrire pour la première fois une étape fondamentale permettant la préparation de l’ADN paternel pour ce voyage périlleux. Ils montrent également que l’activation, anormale, de cette même étape dans des cellules non-germinales (somatiques) est à la base d’un mécanisme oncogénique dans un cancer très agressif. Cette étude a été publiée le 25 Septembre 2018 dans la revue Cell Reports.
Les spermatozoïdes sont les seules cellules capables non seulement de survivre dans un environnement hostile mais aussi de délivrer à l’œuf les gènes du père dans un état d’intégrité parfaite. Avant d’être transporté, cet ADN est préparé par une réorganisation globale et une compaction extrême, lors d’une succession d’évènements sans précédent dans d’autres types cellulaires. Paradoxalement, malgré son importance primordiale pour la procréation et la perpétuation des espèces, ce processus reste l’un des phénomènes les plus obscurs en biologie, bien que des travaux antérieurs aient déjà permis de comprendre dans ses grandes lignes, les mécanismes moléculaires qui dirigent cette compaction du génome mâle.
Un nouveau pas dans la compréhension de ce phénomène a été franchi par les chercheurs en identifiant le mécanisme déclencheur de la compaction de l’ADN mâle et de la métamorphose organisationnelle qui lui est associée. Il faut savoir qu'avant cette compaction, dans les cellules germinales comme dans toutes les cellules de l’organisme, le génome est structuré par des protéines particulières, les histones. Lors de la production des spermatozoïdes, la compaction de l’ADN nécessite l’enlèvement et le remplacement des histones par d’autres protéines, spécifiques des cellules germinales, nommées protamines. On savait précédemment que cet échange histone-protamine était précédé par une modification chimique (acétylation) massive et globale des histones. Or, ni la cause ni la conséquence de cette acétylation massive des histones n’étaient connues.
En suivant la piste initiée par la découverte d’une protéine spécifique de la lignée germinale mâle, de fonction inconnue, les chercheurs ont réussi à identifier le mécanisme de déclenchement de cette acétylation massive des histones et à comprendre l’importance de cette acétylation dans l’enlèvement des histones. Ils montrent que ce même mécanisme, naturellement mis en jeu dans le contexte spécifique de la maturation des cellules germinales mâles, peut aussi être activé de manière aberrante suite à un remaniement génétique et expliquer l’apparition et l’agressivité particulière de certains cancers.
La découverte de ce mécanisme permet non seulement de mieux comprendre une étape essentielle de la vie et de la procréation, mais aussi met en lumière la capacité de certains cancers d’activer des mécanismes moléculaires normaux et naturels de manière hors-contexte pour acquérir des propriétés particulières.
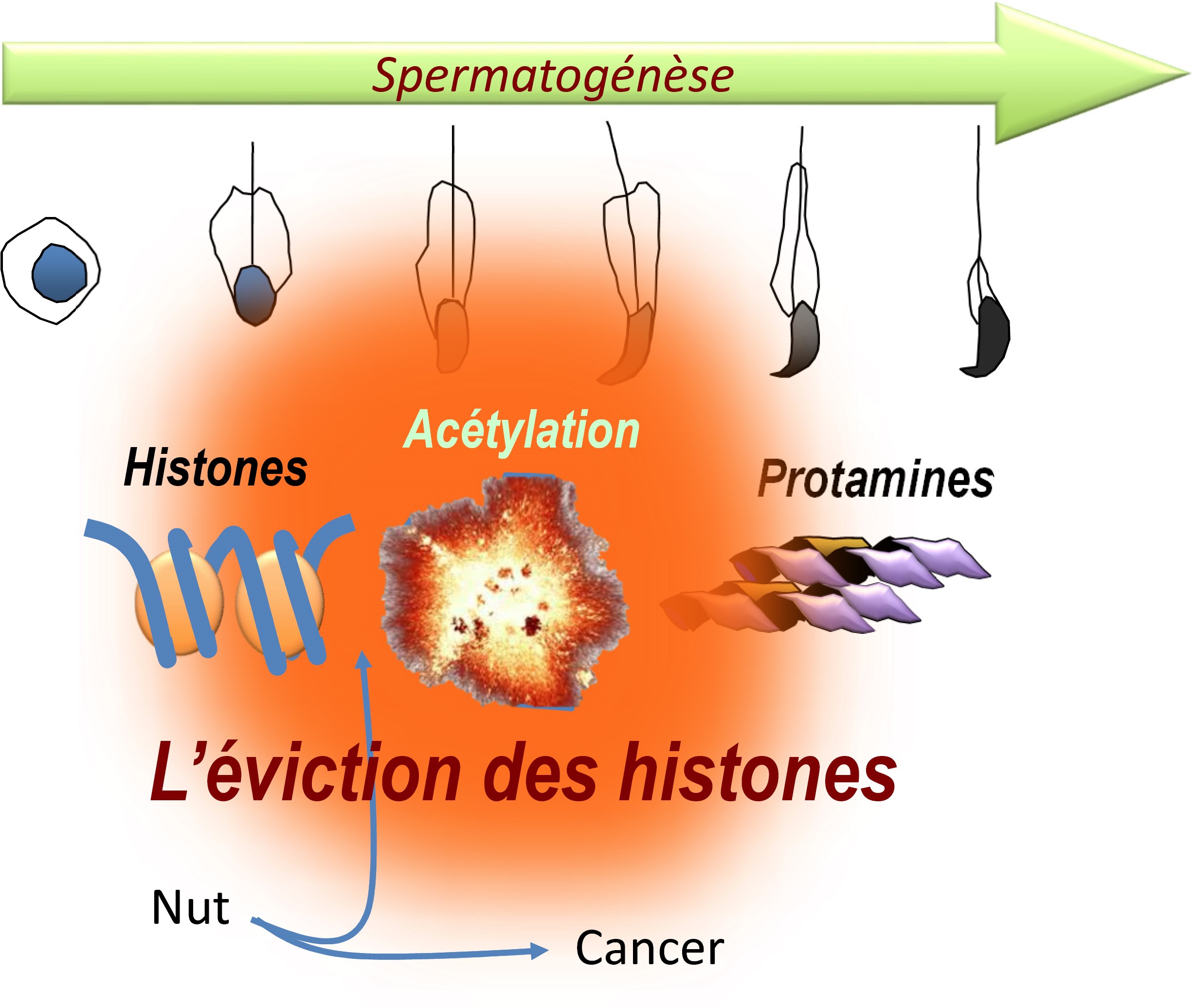
Lorsque ce même facteur Nut est exprimé de manière aberrante, il active des mécanismes similaires à ceux mis en jeu dans son contexte naturel, et ainsi déclenche une acétylation des histones. Cette acétylation non-programmée des histones dans ces cellules non-germinales est à l’origine d’un cancer très agressif.
© Saadi Khochbin
Pour en savoir plus :
Nut Directs p300-Dependent, Genome-Wide H4 Hyperacetylation in Male Germ Cells.
Shiota H, Barral S, Buchou T, Tan M, Couté Y, Charbonnier G, Reynoird N, Boussouar F, Gérard M, Zhu M, Bargier L, Puthier D, Chuffart F, Bourova-Flin E, Picaud S, Filippakopoulos P, Goudarzi A, Ibrahim Z, Panne D, Rousseaux S, Zhao Y, Khochbin S.
Cell Rep. 2018 Sep 25;24(13):3477-3487.e6. doi: 10.1016/j.celrep.2018.08.069.