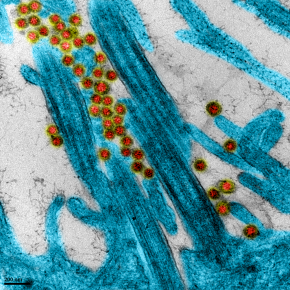
Ces projets qui ont bénéficié de financements en accéléré
Le CNRS a débloqué des fonds très rapidement pour soutenir cinq projets dont les recherches portent sur le COVID-19. À Lille, Lyon, Marseille, Montpellier et Paris, le travail scientifique s’accélère.
Dans la course contre la montre engagée contre le coronavirus, comment accompagner les chercheurs mobilisés à l’avant-poste depuis plusieurs semaines ? « Rapidement, nous nous sommes concertés pour identifier les projets de recherche travaillant sur le SARS-CoV-2 ou les laboratoires disposant déjà des capacités pour le faire » explique André Le Bivic, directeur de l’Institut des sciences biologiques (INSB) du CNRS, qui a travaillé avec Alain Schuhl, directeur général délégué à la science, les délégations régionales du CNRS et les directeurs adjoints scientifiques de l'Institut. L’objectif : faire avancer sans délai des pistes de recherche prometteuses. Ces fonds, d'un montant global de près de 350 000 €, sont ainsi parvenus à cinq équipes en amont des financements issus d'appels à projets.
Repositionner des médicaments
À Lyon, au Centre international de recherche en infectiologie (CIRI)1 , l’équipe VirPath s’est entre autres spécialisée depuis une dizaine d’années dans le repositionnement de médicaments pour le traitement des infections respiratoires. « Nous recherchons aujourd’hui dans la pharmacopée existante ou du côté de molécules « oubliées » si certaines sont susceptibles d’être efficaces contre ce coronavirus » indique Olivier Terrier, chercheur CNRS au CIRI. Grâce au criblage moléculaire, une technique qui consiste à la fois à reproduire et miniaturiser un test biologique, et à l'utiliser pour tester un grand nombre de molécules, « le repositionnement de médicaments permet de gagner un temps précieux pour le développement de traitements efficaces. » ajoute-t-il. Dès janvier dernier, l’équipe a reçu les premiers échantillons issus de patients atteints du COVID-19. Ils ont pu alors caractériser des souches du nouveau coronavirus puis créer des banques virales de travail afin de mettre en place rapidement des modèles précliniques2 . « Nous n’avions évidemment pas de fonds propres à consacrer spécifiquement au COVID-19. Les manipulations en laboratoire de confinement P33 sont très coûteuses » souligne le chercheur. Le soutien du CNRS a donc été bienvenu notamment pour acquérir une chimiothèque complémentaire pour étendre leurs investigations. Aujourd’hui, plus de 55 stratégies de traitements différentes ont déjà été évaluées de manière poussée par l’équipe dont certaines semblent montrer des résultats encourageants.

Ces travaux sont complémentaires à ceux de l’équipe de Jean Dubuisson, directeur de recherche CNRS au Centre d'infection et d'immunité de Lille4 , qui étudie les virus de l’hépatite C, l’hépatite E et les coronavirus depuis 26 ans. « En 2013, lors de l’épidémie de MERS, le syndrome respiratoire du Moyen-Orient, nous avons eu deux cas à Lille. Nous avons alors initié un projet de recherche sur les coronavirus humains pathogènes. » explique le chercheur. Ces financements accélérés du CNRS leur ont permis de faire revenir une post-doctorante pour un an afin de renforcer l’équipe. « Nous avons dû réorganiser l’activité du laboratoire. Trois personnes ont été réaffectées, ce qui donne une force de frappe relativement importante. Nous avons également réorienté notre projet, temporairement, pour faire du criblage de molécules utilisables rapidement en clinique » ajoute-il. « Notre objectif est d’identifier d’ici fin avril des molécules prometteuses pour pouvoir lancer des essais cliniques ».
Mieux comprendre le virus
À Marseille, Bruno Canard5 , directeur de recherche CNRS au laboratoire Architecture et fonction des macromolécules biologiques6 , travaille sur le coronavirus depuis l’épidémie du SARS (syndrome respiratoire aigu sévère), en 2003. Avec son équipe, ils tentent de comprendre comment les enzymes de réplication de l’ARN7 , cibles potentielles pour la mise au point de médicaments antiviraux, travaillent ensemble à fabriquer de nouveaux génomes tout en déjouant les défenses immunitaires de la cellule. « Les deux virus SARS de 2003 et 2019 présentent en effet une similitude quasi parfaite dans leur machine réplicative, c’est-à-dire la machinerie qui copie et reproduit le génome du coronavirus. Si on comprend comment elle fonctionne, nous serons capables de développer des médicaments actifs sur un spectre large de coronavirus précise-t-il. C’est un travail de longue haleine, dont le Remdesivir et l’EIDD-2801 sont une illustration récente ». Ces deux molécules, qui ont déjà montré une activité antivirale contre une grande variété de virus, dont les virus SARS-CoV et MERS-CoV, sont actuellement à l’étude dans certaines chimiothèques. « Il y a encore quelques années, nous étions la seule équipe, en France et en Europe, en pointe sur la biologie structurale des coronavirus. Mais l’assèchement progressif des crédits a considérablement ralenti notre avance. Ces nouveaux financements sont, pour nous, un bol d’air » souligne-t-il. Ces sommes participeront à l’achat d’équipements et de matériel d'aide à la production de protéines du SARS-CoV-2. Leur but : accélérer les connaissances sur la dynamique particulière du virus. Pour lui, « s’il reste beaucoup à découvrir, une brèche s’est ouverte ».
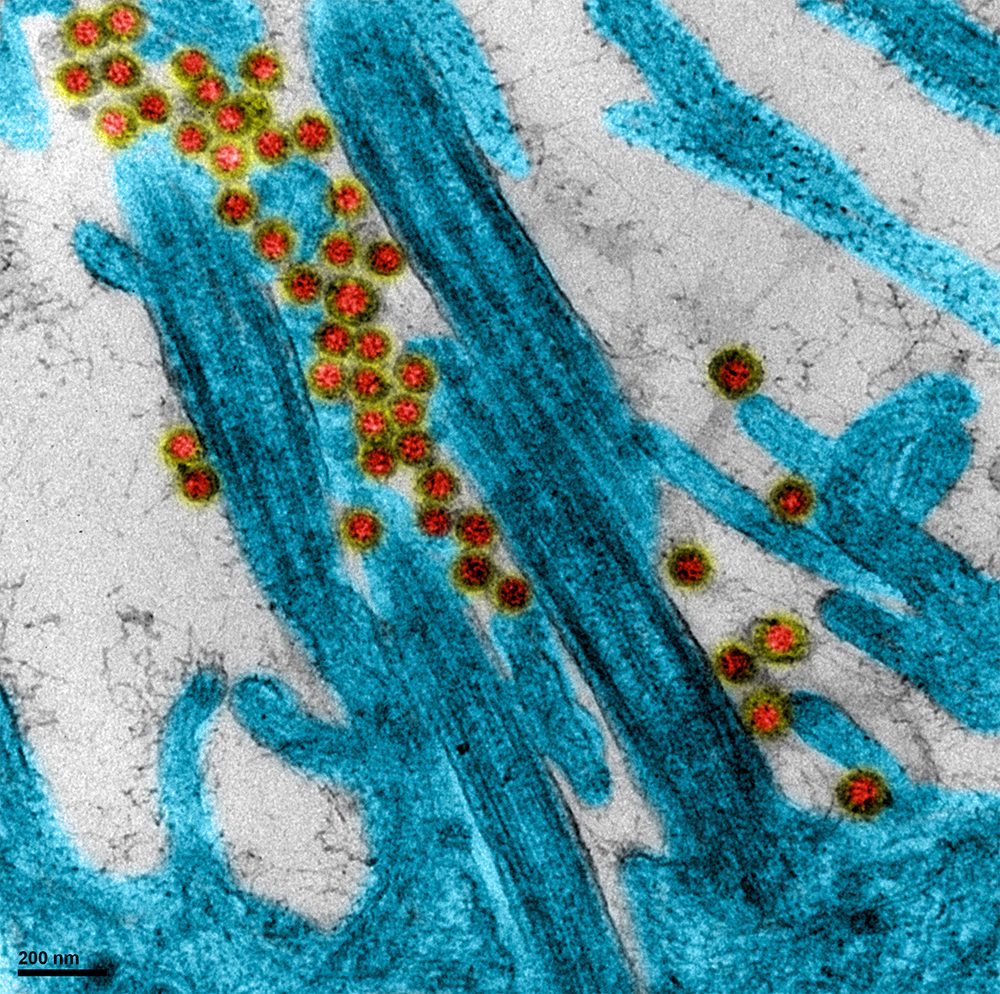
De ses propres mots, c’est par un concours de circonstances que Raphaël Gaudin, chercheur à Institut de recherche en infectiologie de Montpellier8 , s’est confronté à ce nouveau coronavirus. « Au départ, je m’intéressais essentiellement aux interactions hôte-virus, notamment sur les moyens d’empêcher le virus d’entrer dans la cellule » raconte-t-il. En 2016, il publie un article sur l'efficacité d'un antiviral à large spectre sur certains virus enveloppés. « Nous avons poursuivi ces travaux dans mon laboratoire, à l’IRIM, où nous travaillions alors sur le VIH, le virus Zika, les virus de l'hépatites B et C » poursuit le chercheur. Dès janvier, son équipe décide d’inclure le SARS-CoV-2 dans leur protocole. Et depuis quelques semaines, ils se consacrent à plein temps au développement de nouvelles molécules antivirales. « Grâce au soutien du CNRS, nous avons pu financer une partie du matériel nécessaire et spécifique au coronavirus. C’est une véritable opportunité pour nous de pouvoir avancer plus vite. » déclare-t-il. En plus de nouveaux équipements, ces moyens exceptionnels seront utilisés pour accélérer la synthèse de molécules afin de pouvoir les améliorer davantage avant d’enclencher des études in vivo. Dans la foulée, d’autres équipes se sont jointes à l’effort : à l’IRIM, un consortium s’est constitué pour pouvoir travailler, en synergie, sur le COVID-19.
Une production à l’échelle industrielle
À l’Institut de biologie de l’École normale supérieure9 , Lionel Navarro, chercheur CNRS et spécialiste des interactions hôtes-bactéries pathogènes, et son équipe étudient un mécanisme de régulation génique via des petits ARN dont l’activité antivirale est déjà bien établie. Ces dernières années, l’équipe a pu montrer qu’il était possible de cibler à façon, et de manière sélective, des gènes de virulence ou de survie de bactéries pathogènes et ainsi bloquer leur expression. En collaboration avec une spin-off de l’IBENS, ImmunRise, les chercheurs travaillent actuellement au développement d’un système simple de production de petits ARN antimicrobiens. « Ce système biologique est adapté à la production à grande échelle et industrialisable. Le génome du SARS-CoV-2 étant connu, nous envisageons d’exploiter ce système pour produire rapidement des petits ARN dirigés contre 12 régions du génome viral » explique Lionel Navarro. Pour les scientifiques de l’IBENS, les financements du CNRS ont été essentiels afin d’amorcer le projet. « Nous avons pu acquérir deux photobioréacteurs, afin de produire suffisamment de petits ARN vectorisés et tester ainsi leur efficacité antivirale dans les cellules humaines et de souris10 . Nous espérons que ce projet collaboratif donnera un coup d’accélérateur au processus de maturation de notre technologie » ajoute le chercheur. Et d'apporter une nouvelle stratégie de lutte contre le COVID-19 qui pourra être exploitée ultérieurement contre d'autres maladies émergentes.
En soutenant la recherche sur tous les fronts, le CNRS réaffirme la nécessaire constitution d’une première ligne de défense scientifique. « Il s’agit également pour nous d’évaluer si nos laboratoires et nos scientifiques sont préparés et bien armés pour faire face à cette crise sanitaire sans précédent et pour contribuer à freiner, puis stopper la pandémie. Car on ne sait pas d’où viendra la prochaine menace. » conclut André Le Bivic.
- 1(CNRS/Inserm/ENS Lyon/Université Claude Bernard Lyon 1).
- 2À partir de tissus humains constitués d’épithélium respiratoire reconstitué et cultivé en interface air/liquide, très proches de la physiologie in vivo.
- 3Dans un laboratoire, le confinement vise à isoler des organismes pathogènes (bactéries, virus, prions ou parasites) dans une zone aménagée spécifiquement et techniquement de sorte que le personnel soit protégé d'un risque biologique ou chimique. Il existe trois niveaux de confinement (P2, P3 et P4) correspondant respectivement à l’utilisation d’agents biologiques des groupes 2, 3 et 4. Le niveau de confinement 3 est constitué de zones à atmosphères contrôlées et convient à la manipulation des agents pathogènes provoquant généralement une maladie humaine grave voire mortelle, ou ayant de lourdes conséquences économiques, transmissibles par voie aérienne.
- 4CIIL (INSERM/CNRS/Université de Lille/Institut Pasteur de Lille/CHU de Lille).
- 5À lire, son interview dans CNRS le Journal https://lejournal.cnrs.fr/articles/la-science-fondamentale-est-notre-meilleure-assurance-contre-les-epidemies
- 6CNRS/Aix-Marseille Université/INRAE.
- 7Présent chez tous les êtres vivants, l’ARN (acide ribonucléique) se retrouve chez certains virus. Si l’ADN (acide desoxyribonucléïque) est le support de l’hérédité, l’ARN, molécule fabriquée sur le modèle de l’ADN, est essentiel dans le transport du message génétique et la synthèse des protéines.
- 8IRIM (CNRS/Université de Montpellier).
- 9IBENS (CNRS/ENS/Inserm).
- 10Cette seconde étape sera normalement réalisée à l'Institut Pasteur, en collaboration avec l'équipe de Marco Vignuzzi.